10000種類以上のmRNAをsubcellular resolutionで解析できそうなSpatial transcriptomicsの手法について、宮脇が5つの原理に分けて簡単にレビュー。
From Alon et al., bioRxiv 2020.05.13.094268 (2020)
Show Notes:
1. RNAのcDNAを環状にして増幅、SOLiDでin situ sequencing
- Highly Multiplexed Subcellular RNA Sequencing in Situ…FISSEQ。George Churchラボ。
- Rolling Circle Amplification…環状にした一本鎖DNAテンプレートに対して一方向にポリメラーゼで増幅をかけ、何周もさせることにより、巨大なアンプリコンを作成する手法
- Next Gen SOLiD DNA Sequencing Method Explained…SOLiDの原理説明動画。In Situ Sequencingの文脈で関連があるのは 5:18~。
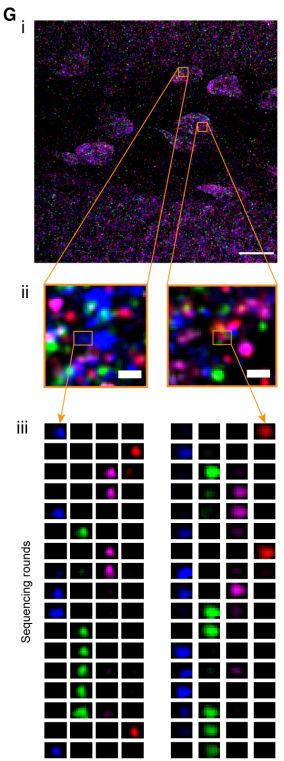
- Expansion sequencing: Spatially precise in situ transcriptomics in intact biological systems…UntargetedとTargetedの2種類があるが、前者はFISSEQ+ExM。Boyden+Churchラボ。
- Expansion microscopy…脳サンプルを大きくすることにより実効的な解像度を上げる手法
2. Padlock ProbeでmRNAとDNAバーコードを対応付けてin situ sequencing
- Padlock Probe…末端の配列が、隣接する標的配列を持つオリゴヌクレオチド。標的配列にハイブリダイゼーションすると”ブリッジ”するような形になり、ライゲーションによって環状化できる。
- In situ sequencing for RNA analysis in preserved tissue and cells…Padlock Probeを作ったMats Nillsonたちによるin situ sequencing
- SplintR® Ligase…これのお陰でPadlockを使う際の逆転写が不要になった。PBCV-1 DNA ligaseとも。
- Expansion Sequencing…Targetedはこちらの原理を採用。
- Three-dimensional intact-tissue sequencing of single-cell transcriptional states…DeisserothラボのSTARmap。PadlockやSOLiDについて改良しているけど、この分類に入れちゃいます。
- Barcoded oligonucleotides ligated on RNA amplified for multiplexed and parallel in situ analyses…BOLORAMIS。George Churchラボ。
- High-Throughput Mapping of Long-Range Neuronal Projection Using In Situ Sequencing…BARseq。Zadorラボ。MAPseqによって既に細胞に発現させているRNAのバーコードをpadlockに取り込み、増幅。内在性のmRNAも一緒に読める。bioRxivにある改良版もチェック。
- High-Throughput Mapping of Single-Neuron Projections by Sequencing of Barcoded RNA..MAPseq。Zadorラボ。Sindbis Virusを使って核酸のバーコードを、それを軸索まで運ぶための人工タンパク質と一緒に細胞に導入する。ある脳領域にウイルスを局所注入し、投射先領域の切片からRNAシーケンシングを行ってどのバーコードが存在していたかを調べれば、個々のニューロンがどこに投射していたのかを一挙に知ることができる。
3. 最初にhybridizeするプローブにバーコードの組み合わせを持たせ、更にバーコードに対するhybridizationを複数回行う
- Researchat #54…同じく生物学系ポッドキャスト。SeqFISH,MERFISHあたりの技術がわかりやすく解説されている。
- Spatially resolved, highly multiplexed RNA profiling in single cells…MERFISH。Xiaowei Zhuangラボ。
- Transcriptome-scale super-resolved imaging in tissues by RNA seqFISH…seqFISH plus。Long Caiラボ。このラボは他のin situ sequence手法もたくさん出しているので気になる方はラボHPをチェック。訂正:seqFISH Plusの組み合わせ数の説明を間違えました…。すみません。一つの遺伝子に用いる色は1色のみ、プローブについた4スロットのバーコードは独立したラウンド(1ラウンドあたり20回のハイブリダイゼーション)で染色、1スロットはError Correction用、ということで、分離できるのは60C4個ではなく3×20^(4-1)個。
- smFISH…一つのmRNAに対して何個もハイブリダイズプローブを結合させることによりシグナル増幅を行う手法
4. 座標ごとにバーコードをつけたスライドガラスに組織を貼り付ける
- Visualization and analysis of gene expression in tissue sections by spatial transcriptomics…Spatial Transcriptomics. Frisenラボ。1stのStahlは独立して更に手法を進化させている。
- Slide-seq: A scalable technology for measuring genome-wide expression at high spatial resolution…Fei Chen+Evan Macosko。Drop-seqの技術を応用。解像度10 um。
- Large field of view-spatially resolved transcriptomics at nanoscale resolution…Stereo-Seq
- Seq-Scope: Submicrometer-resolution spatial transcriptomics for single cell and subcellular studies…Seq-Scope
- Continuous Polony Gels for Tissue Mapping with High Resolution and RNA Capture Efficiency…PIXEL-Seq
5. mRNA同士の近さをDNAで記録し、距離行列を次元削減
- Researchat #16…DNA microscopy回。
- DNA Microscopy: Optics-free Spatio-genetic Imaging by a Stand-Alone Chemical Reaction…Aviv Regev, Feng Zhangラボから。Overhang PCRでmRNAの距離情報をPCR産物として記録。筆頭著者のJoshu Weinsteinによるプレゼンも分かり易い。
- A DNA nanoscope via auto-cycling proximity recording…DNA nanoscope from Peng Yinラボ。mRNAを対象としているわけでは無いけど、今後応用できるかもねってことで。Auto-cycling proximity recordingというギミックを利用。
- Nature Reviews drug discoveryによる直近のAviv Regevのインタビュー記事。
その他
- Expansion-Assisted Iterative-FISH defines lateral hypothalamus spatio-molecular organization…EASI-FISH from Sternson/Tilbergラボ。みんなができるSpatial Transcriptomeというコンセプトで作られた手法。染色の回数と見ることができるmRNAの種類はリニアな関係にしかないが、高々数10種類のマーカーを見るならこれでいい気もする。300 um厚の切片に適用。
Editorial Notes:
- 一般にSpatial Transcriptomicsというと、今回紹介したものの他に、マイクロダイセクションをベースとしたもの(STRP-seqとか)や、scRNAseqのデータから座標をcomputationalに推定する(Tangramとか)、という方法も含みます。今回はsubcellular resolution担保できなさそう、ってことで割愛(宮脇)
- 当初このネタでポッドキャストをキックオフしようかと思っていましたが、直前にresearchatパイセンの#54を発見し、あまりにもコンセプトが被っていてかつわかりやすかったので、神経科学への応用に寄せて寄せて無事お披露目となりました汗 (萩原)